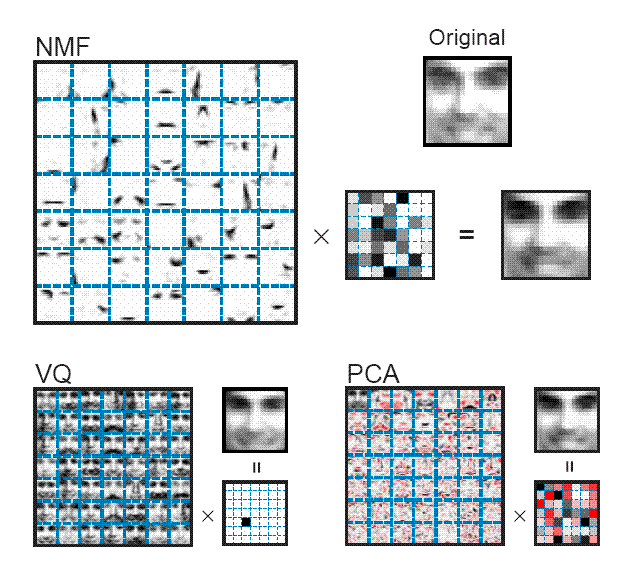
Ekstrakcja cech
Rapid Miner
- Uruchom narzędzie RapidMiner
- Utwórz nowy przepływ i umieść w nim zbiór danych winequality-red.csv. Pamiętaj, że procedura PCA jest zależna od skal atrybutów, więc w celu uniknięcia błędu dokonaj standaryzacji atrybutów numerycznych (operator
Normalizez opcją transformacjiZ-transform). Zaaplikuj do zbioru danych operatorPrincipal Component Analysisi postaraj się zachować 95% oryginalnej wariancji zbioru danych. Do ilu cech udało się ograniczyć zbiór danych? Czy potrafisz zinterpretować uzyskane cechy? - Czy sposób normalizacji wpływa na uzyskiwane wyniki? Spróbuj rzutować wszystkie atrybuty do zakresu <0,1> i sprawdź kumulatywną tłumaczoną wariancję.
- Wykorzystaj plotter Scatter do wizualizacji uzyskanego modelu, ograniczając się do pierwszych trzech składowych głównych.
- Uznała(e)ś, że jakość analizy nie ulegnie znaczącemu pogorszeniu jeśli zdecydujemy się na utratę do 50% oryginalnej wariancji zbioru danych. Ile teraz uzyskała(e)ś cech? Jakie wnioski można wyciągnąć z wizualizacji uzyskanego modelu?
- Zamień operator
Principal Component Analysisna operatorSingular Value Decomposition. Obejrzyj uzyskany wynik. Czym różni się ten wynik od modelu PCA? - Wykorzystaj opreator
Read CSVdo wczytania zbioru danych autos.csv. Wskaż atrybutBODY_STYLEjako zmienną celu. Wykorzystaj któryś z algorytmów ekstrakcji cech do zmniejszenia liczby cech do 3. Dokonaj wizualizacji uzyskanych wyników. Czy możesz rozpoznać w nowej przestrzeni atrybutów poszczególne typy samochodów?
Python
- uruchom notatnik Jupyter obrazujący możliwość wykorzystania PCA i samoorganizujących się map (tzw. map Kohonena).
zadanie samodzielne
Celem zadanie jest przetworzenie zbioru danych cukrzyca.csv. Zbiór posiada następujące atrybuty:
PREGNACIES: liczba wcześniejszych ciążGLUCOSE: koncentracja glukozy w 2 godziny po doustnym podaniu próbki testowejBLOOD_PRESSURE: ciśnienie krwi (mm Hg)SKIN_THICKNESS: grubość fałdu skóry na tricepsie (mm)INSULIN: poziom serum insuliny (mu U/ml)BMI: body mass index (waga w kg podzielona przez (wysokość w m2)DIABETES_PEDIGREE: funkcja występowania cukrzycy w rodzinieAGE: wiekCLASS: atrybut decyzyjny wskazujący na obecność/brak cukrzycy
Postaraj się zastąpić zbiór oryginalnych atrybutów przez mniejszy zbiór cech. Sprawdź, do jakiego stopnia zmniejszenie liczby atrybutów wpływa na dokładność klasyfikacji. Wykonaj ćwiczenie wg poniższego wzorca:
- zbuduj klasyfikator (np. drzewo decyzyjne) wykorzystujący oryginalny zbiór danych, zanotuj dokładność klasyfikatora
- dokonaj ekstrakcji dwóch cech ze zbioru (możesz wykorzystać PCA, SVD, NNMF)
- zastosuj model ekstrakcji cech zbudowany w punkcie (2) do danych, aby przetransformować dane do nowej przestrzeni zdefiniowanej przez odkryte cechy
- zbuduj klasyfikator (wykorzystaj ten sam algorytm co w punkcie (1)) działający na danych z punktu (3), porównaj uzyskaną dokładność klasyfikatora.
UWAGA
- do testowania klasyfikatora użyj 10-krotnej walidacji krzyżowej
- w pliku
*.csvnie ma zdefiniowanych atrybutów, musisz dodać je samodzielnie przed przystąpieniem do analizy - dane są numeryczne, rozważ normalizację atrybutów i odkrywanych cech
- budując model ekstrakcji cech pamiętaj o tym, aby koniecznie wyłączyć z budowy atrybut
CLASS - za realizację zadania można uzyskać:
- dodatkowe 5 XP za uzyskanie największej dokładności na danych w przestrzeni 2D (punktowanych jest pięć najlepszych rozwiązań)
- dodatkowe 5 XP za znalezienie algorytmu klasyfikacji, który najmniej traci dokładności po redukcji wymiarów (punktowanych jest pięć najlepszych rozwiązań)
Rozwiązania proszę wysłać na adres Mikolaj.Morzy@put.poznan.pl w postaci pliku *.pdf zawierającego:
- zrzut ekranu z procesem
- macierz pomyłek dla oryginalnych danych
- macierz pomyłek dla danych w przestrzeni 2D
- informację o dokładności klasyfikacji w oryginalnej przestrzeni i w przestrzeni zredukowanej do 2D
Rozwiązania przyjmuję do piątku, 19 czerwca 2020 r., do godziny 21:00.
Except where otherwise noted, content on this wiki is licensed under the following license:Public Domain